什么是ATAC-seq
Assay for Transposase-Accessible Chromatin with highthroughput sequencing(ATAC-Seq)即利用转座酶探究可接近性染色质高通量测序技术。通俗来说就是利用转座酶来获取开放性染色质,再通过高通量测序及生物信息学分析来挖掘相关基因信息,以此探究生物学相关问题1。ATAC-Seq是MNase-seq,DNase-seq和FAIRE-seq的替代或补充技术,用于染色质可及性分析。从ATAC-seq获得的结果与从DNase-seq和FAIRE-seq获得的结果相似。与其他技术相比,ATAC-seq越来越受欢迎,因为它不需要交联,具有更高的信噪比,需要的生物材料数量少得多且执行起来更快,更容易2。
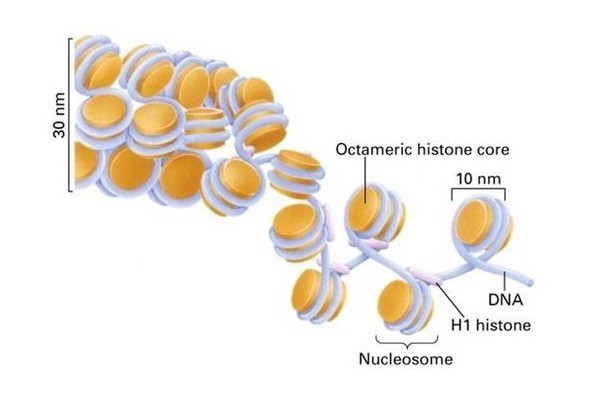
Image from 1
为什么研究染色质开放区域?
染色质分为常染色质和异染色质,在结构上常染色质折叠压缩程度低,处于伸展状态,DNA复制,基因转录都发生在DNA的致密高级结构变为松散的状态;这部分打开的染色质,就叫开放染色质(open chromatin)。而打开的染色质,就有足够的区域允许一些调控蛋白(比如转录因子和辅因子)过来与之相结合。而染色质的这种特性,就叫做染色质的可接近性(chromatin accessibility)。通过研究细胞特定状态下开放的染色质区域可以在DNA水平上了解其转录调控1。
如何寻找开放的染色质区域?
传统使用的的实验方法主要是有MNase-seq和DNase-seq ,这两种实验方法的主要思路是:染色质变得开放,就意味着DNA和组蛋白的聚集程度降低,就会有一部分DNA暴露出来。而一旦失去了蛋白质的保护,这部分DNA就可以被DNA酶(MNase或DNase I)所切割。然后,我们再把切割完的DNA拿来测序,和已知的全基因组序列相比较,就能发现被切割的是哪些序列,没有被切掉的基因序列又在哪里,就知道开放的染色质区域在哪里了。不过,这两个方法有明显的缺陷,即耗时费力与重复性差。虽然FAIRE-seq 不依赖酶和抗体,但其检测背景较高,测序信噪比低,甲醛交联时间不好把握等缺陷,限制其使用范围1。
有什么新技术方法来研究开放染色质?
新推出的ATAC-seq利用Tn5转座酶(DNA转座,是一种把DNA序列从染色体的一个区域搬运到另外一个区域的现象,这一过程就由转座酶参与完成。Tn5转座酶:“标签片段化工具”,Tn5转座体可将其衔接子负载整合到可接近的染色质区域,而空间位阻较不可接近的染色质使得转座不可能发生。)人为将将携带已知DNA序列标签的转座复合物,加入到细胞核中,再利用已知序列的标签进行PCR建库测序,就知道哪些区域是开放染色质了。ATAC-seq出来的结果,和传统方法出来的结果具有很强的一致性,同时也和ChIP-seq有较高的吻合程度。而相比较而言,ATAC-seq的重复性,比MNase-seq和DNase-seq的更强,操作起来也更加简便,而且只需要很少的细胞/组织量,同时测序信号更加好。目前已经成为研究染色质开放性首选的技术方法1。 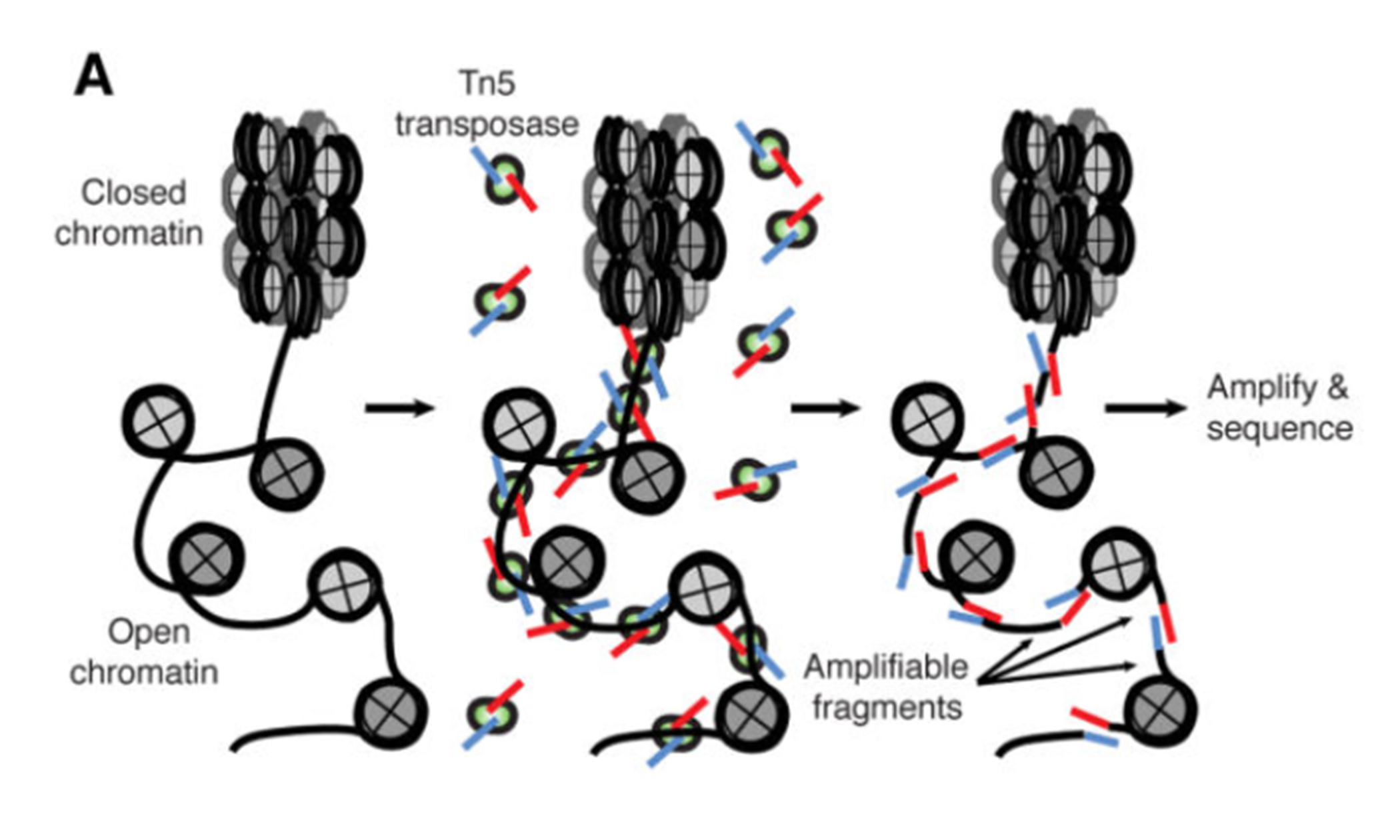 Image from 3
Image from 3
ATAC-seq 流程
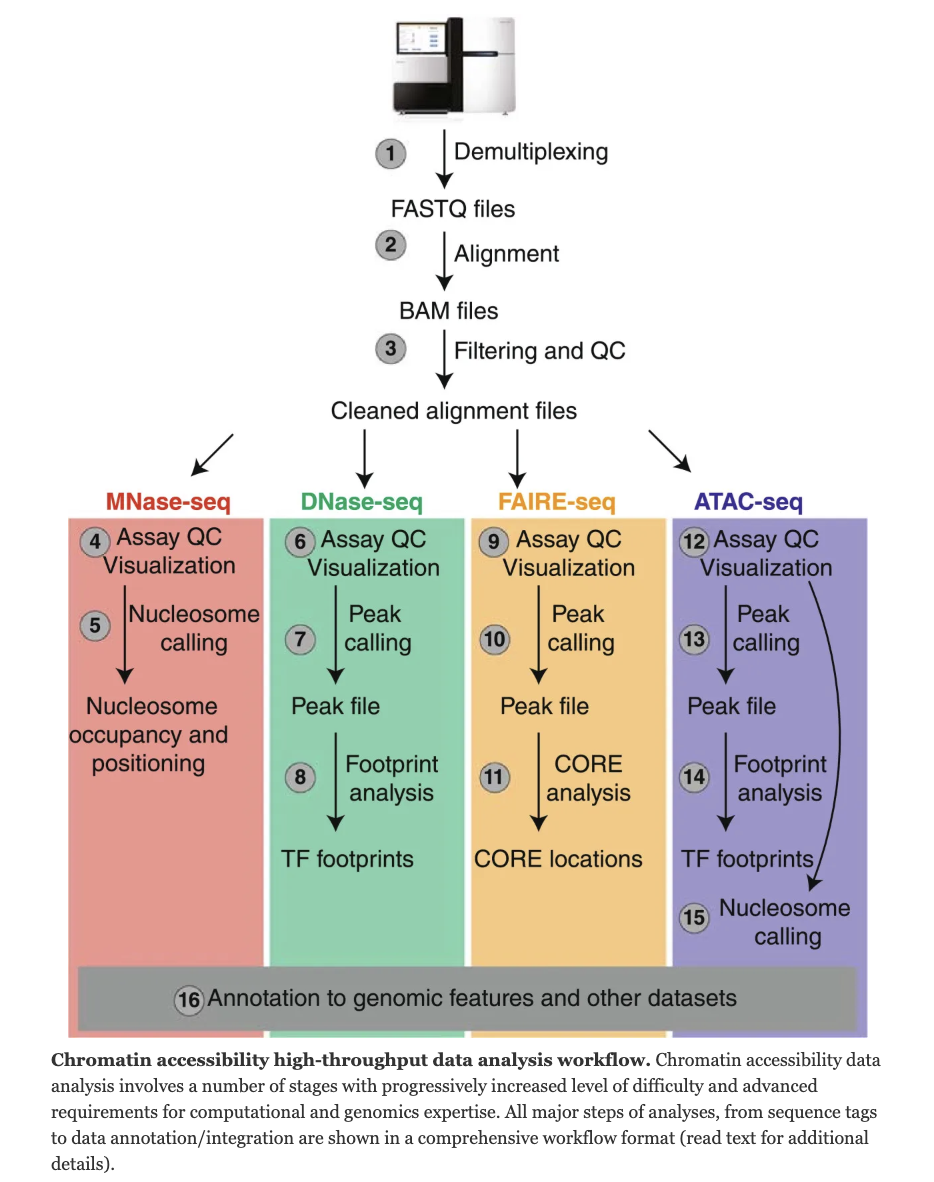 Image from 4
Image from 4
参考
https://bioconductor.org/packages/devel/bioc/vignettes/ATACseqQC/inst/doc/ATACseqQC.html ↩
ATAC-seq: A Method for Assaying Chromatin Accessibility Genome-Wide ↩