转录起始位点TSS
转录起始位点(Transcription Start Site, TSS)是指一个基因的5’端转录的第一个碱基,它是转录时,mRNA链第一个核苷酸相对应DNA链上的碱基,通常是一个嘌呤(A或G)。通常把转录起始点(即5‘末端)前的序列称为上游(upstream),而把其后(即3’末端)的序列称为下游(downstream)。启动子(promoter)包含转录点位,这两者是包含与被包含的关系。启动子是一段DNA片段,一般在转录起始位点的上游,启动子与与RNA聚合酶结合并能够起始mRNA的合成,启动转录1。由于TSS是RNA分子5’转录的起始位点,那么知道了TSS的确切位置就可以立即确定其侧面的调控区2。
在转录起始过程中,RNA聚合酶(RNAP)与启动子DNA结合,解开启动子DNA形成包含单链“转录气泡”的RNAP-启动子起始复合物(PIC),并选择一个转录起始位点(TSS)。根据启动子序列和起始底物浓度(initiating-substrate concentration),TSS选择发生在启动子区域内的不同位置3。
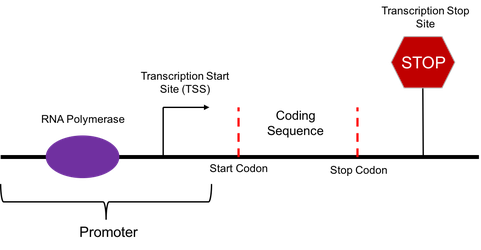 Image from 4
Image from 4
启动子 Promoter
转录起始于DNA模板上的启动子。 启动子是将RNA聚合酶引导至适当的转录起始位点的DNA序列。
核心启动子 Core Promoter
核心启动子区域位于最接近起始密码子(start codon)的位置,包含RNA聚合酶结合位点,TATA框和转录起始位点(TSS)。 RNA聚合酶将稳定地结合/绑定(bind)到该核心启动子区域,并且启动模板链的转录。 TATA盒是核心启动子区域内的DNA序列(5’-TATAAA-3’),一般转录因子蛋白和组蛋白可以bind在该区域。 组蛋白(Histones)是在真核细胞中发现的将DNA包装成核小体的蛋白质。 组蛋白的bind会阻止转录的启动,而转录因子促进转录的启动。 核心启动子的最3’部分(最接近基因的起始密码子)是TSS,它实际上是转录开始的地方。 但是,只有真核生物和古细菌包含此TATA盒。 大多数原核生物包含一个被认为功能上等效的序列,称为Pribnow盒,通常由六个核苷酸TATAAT组成4。
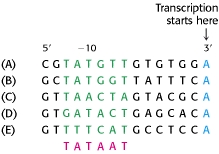 Image from 5
Image from 5
当比较许多原核启动子的序列时,在起始位点的5’(上游)侧存在两个常见的motif。 它们被称为-10序列和-35序列,因为它们位于起始位点上游约10和35个核苷酸的中心。
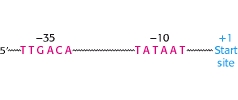 Image from 5
Image from 5
近端启动子 Proximal Promoter
近端启动子在核心启动子的上游,包含许多主要调控元件。 在TSS上游约250个碱基对处发现了近端启动子,它是一般转录因子结合的位点4。
远端启动子 Distal Promoter
启动子区域的最后部分称为远端启动子,它位于近端启动子的上游。 远端启动子也包含转录因子结合位点,但主要包含调节元件4。
染色质和染色体
每一条染色单体由单个线性DNA分子组成。细胞核中的DNA是经过高度有序的包装(或盘绕),否则就是一团乱麻,不利于DNA复制和表达调控。这种有序的状态才能保证基因组的复制和表达调控能准确和高效进行。
包装分为多个水平,核小体核心颗粒(nucleosome core particle)、染色小体(chromatosome)、 30 nm水平染色质纤丝(30 nm fibre)和高于30 nm水平的染色体包装。在细胞周期的不同时期,DNA的浓缩程度不同,细胞分裂间期表现为染色质具有转录活性,而中期染色体是转录惰性。细胞主要处于分裂间期,所以DNA大部分时间都是染色质而不是染色体,只不过大家喜欢用染色体泛指染色质和染色体6。
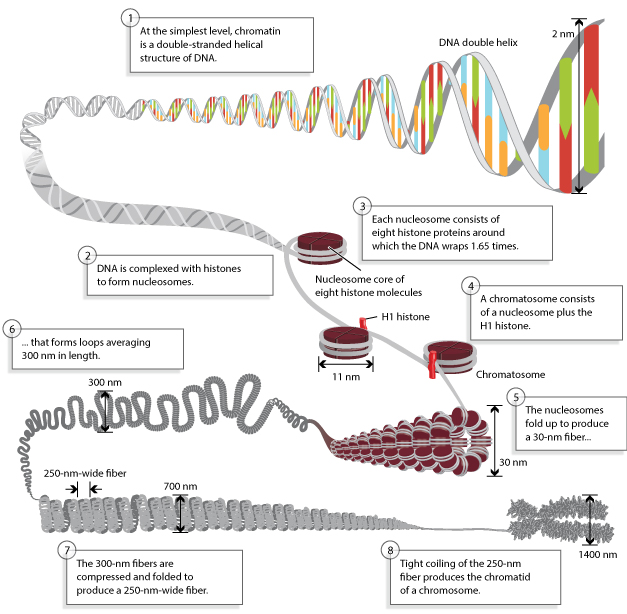 Image from 7
Image from 7
核小体 Nucleosome 与 无核小体区域 NFR
核小体是染色质的基本结构单位,由DNA和H1、H2A、H2B、H3和H4等5种组蛋白(histone,H)构成。两分子的H2A、H2B、H3和H4形成一个组蛋白八聚体,约200 bp的DNA分子盘绕或包装在组蛋白八聚体构成的核心结构外面1.75圈形成了一个核小体的核心颗粒(core particle)。在这 200bp中,146 bp是直接盘绕在组蛋白八聚体核心外面,这些DNA不易被核酸酶消化,其余的DNA(约60bp)和组蛋白H1共同构成的连接区(linker)用于连接下一个核小体,于是形成串珠状的染色质细丝8,9, 这是染色质的主要且最稳定的结构水平。有丝分裂时染色质进一步压缩为染色体。
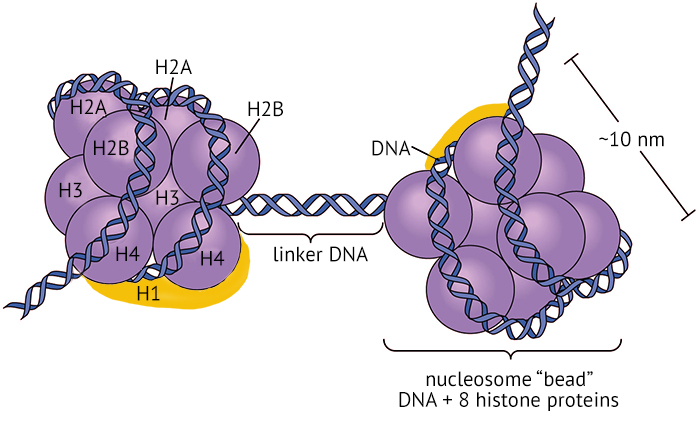 Image from 10
Image from 10
染色质的核小体与转录调控相关。用内切核糖酶–微球菌核酸酶(micrococcal nuclease, MNase, MN酶)处理染色质可以得到单个核小体。DNA暴露在核小体表面使得其能被特定的核酸酶接近并切割。染色质结构改变会发生在与转录起始相关或与DNA的某种结构特征相关的特定位点。当染色质用DNA酶I(DNase)消化时,第一个效果就是在双链体中特定的超敏位点(hypersenitive site)引入缺口,这种敏感性可以反应染色质中DNA的可及性(accessible),也就是说这些是染色质中DNA由于未组装成通常核小体结构而特别暴露出的结构(开放区域)。许多超敏位点与基因表达有关,每个活性基因在启动子区域都存在一个超敏位点。大部分超敏位点仅存在于相关基因正在被表达的或正在准备表达的细胞染色中;基因表达不活跃时他们则不出现6。
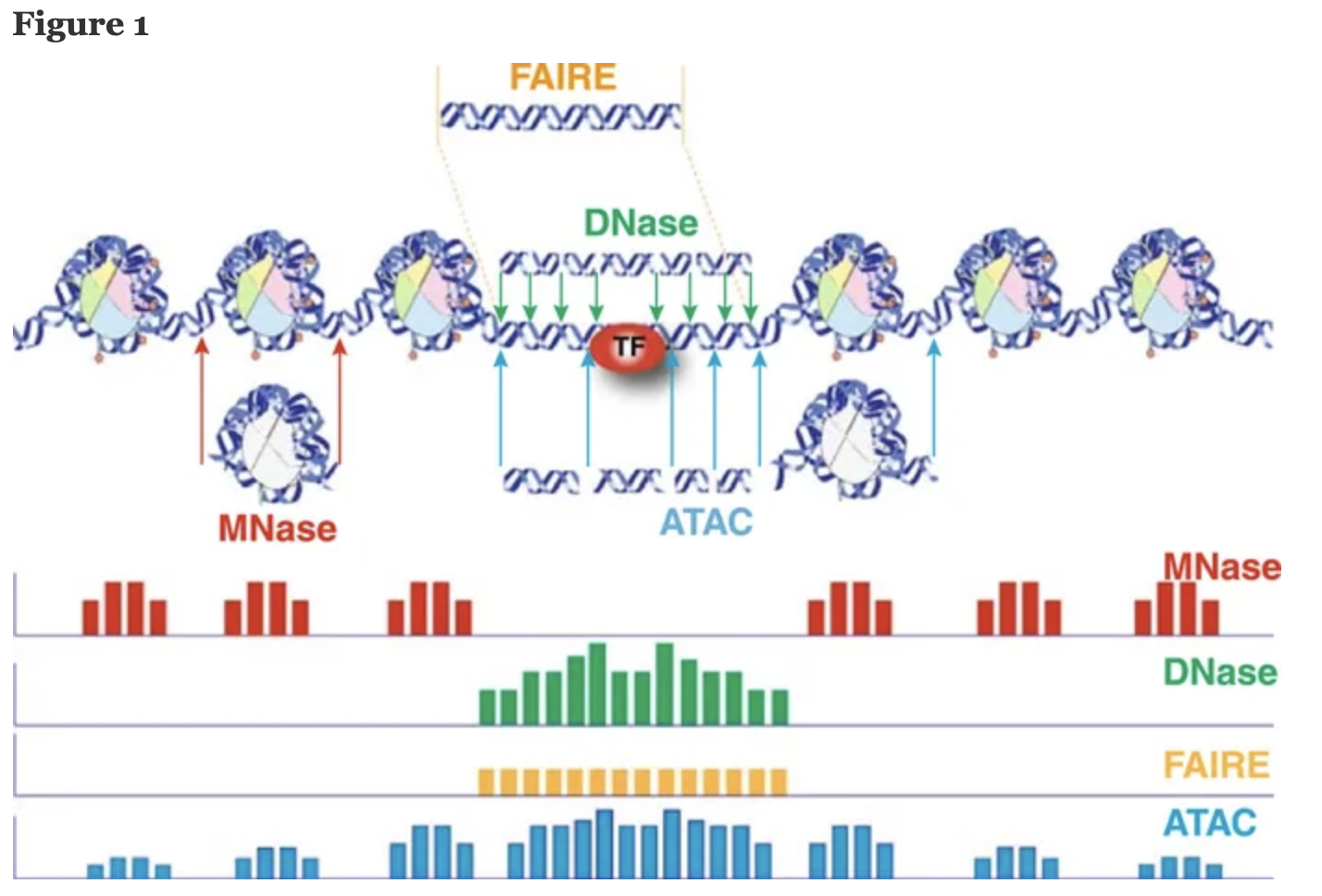 Image from 11
Image from 11
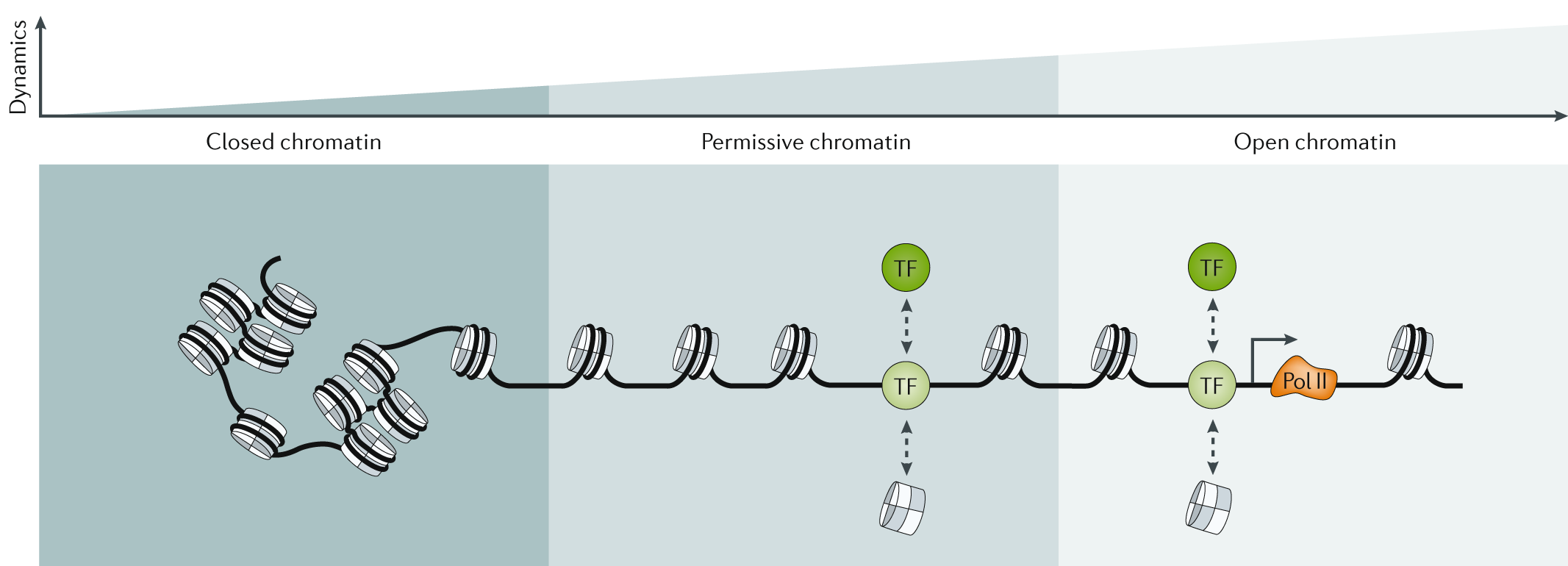 Image from 12
Image from 12
超敏位点和基因表达有关,并且超敏位点反应了染色质的可及性。也就可以反推出“可及性”的染色质结构区域可能与基因表达调控相关。于是2015年的一篇文章Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNDNA-binding proteins and nucleosome position就使用了超敏Tn5转座酶切割染色质的开放区域,并且加上接头(adapter)进行高通量测序。
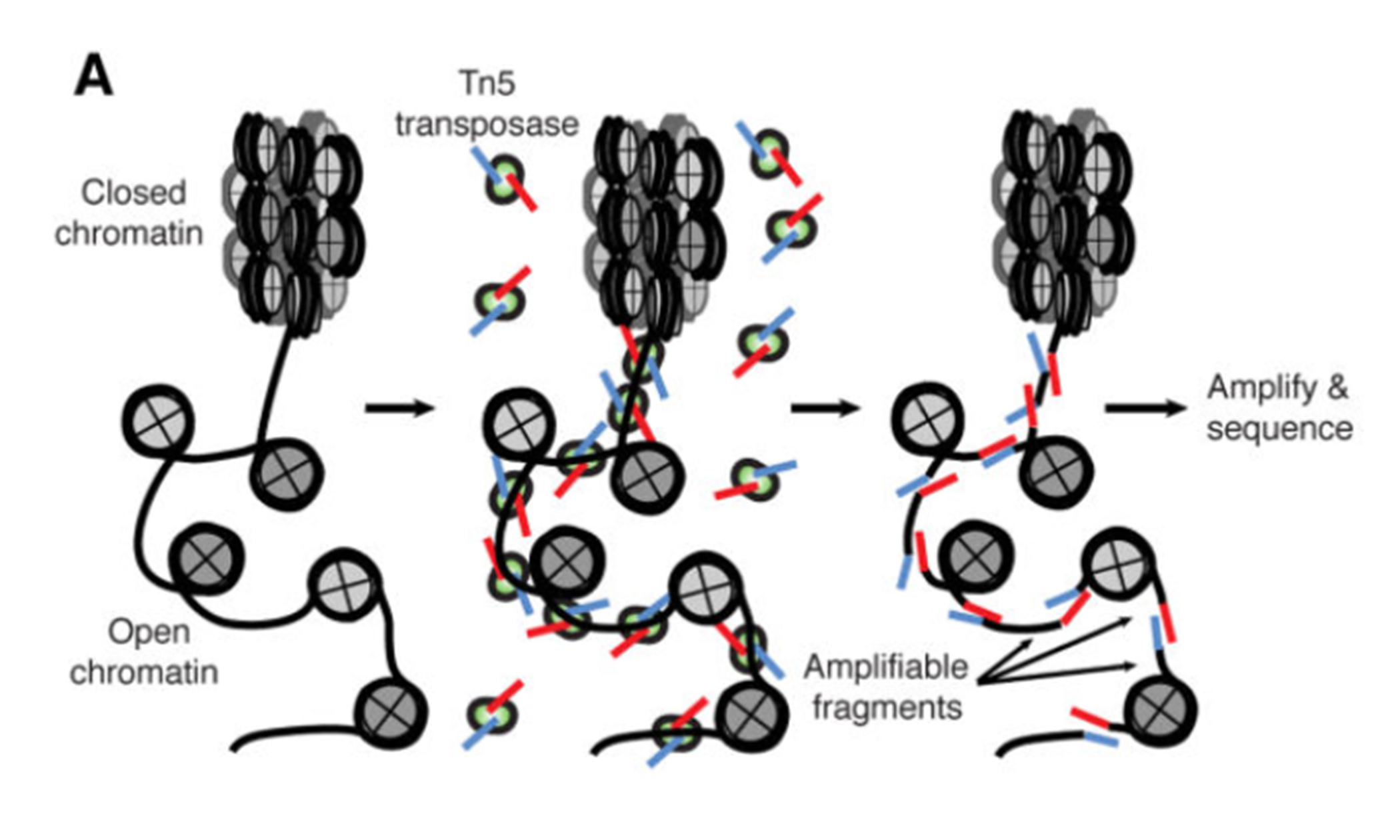 Image from 13
Image from 13
真核生物(eukaryotic)基因组中核小体的位置决定了调节蛋白容易进入DNA序列的哪些部分,而哪些则不容易。核小体位置的全基因组图谱揭示了转录起始位点TSS附近(特别注意启动子区域)涉及一个无核小体区域(NFR)的显著模式:即无核小体区域侧翼的平均核小体密度表现出的明显相邻振荡的周期性模式8,14, 这是由于核小体经常启动子经常耗尽14。
参考文献:
https://www.encodeproject.org/data-standards/terms/#enrichment
Kapranov, Philipp. “From transcription start site to cell biology.” Genome biology 10.4 (2009): 217. ↩
The mechanism of variability in transcription start site selection ↩
https://biology.stackexchange.com/questions/58648/is-the-transcription-starting-site-located-before-or-after-the-promoter ↩ ↩2
Luger, Karolin, et al. “Crystal structure of the nucleosome core particle at 2.8 Å resolution.” Nature 389.6648 (1997): 251-260. ↩ ↩2
https://baike.baidu.com/item/%E6%A0%B8%E5%B0%8F%E4%BD%93 ↩
https://www.philpoteducation.com/mod/book/view.php?id=802&chapterid=1072#/ ↩
ATAC-seq: A Method for Assaying Chromatin Accessibility Genome-Wide ↩
Lee, William, et al. “A high-resolution atlas of nucleosome occupancy in yeast.” Nature genetics 39.10 (2007): 1235-1244. ↩ ↩2