基于参考基因组查找给定物种所有已标注基因的TSS
这里参考生信菜鸟团的方法。
1. 首先在UCSC的 table browser 设置并下载text文件:
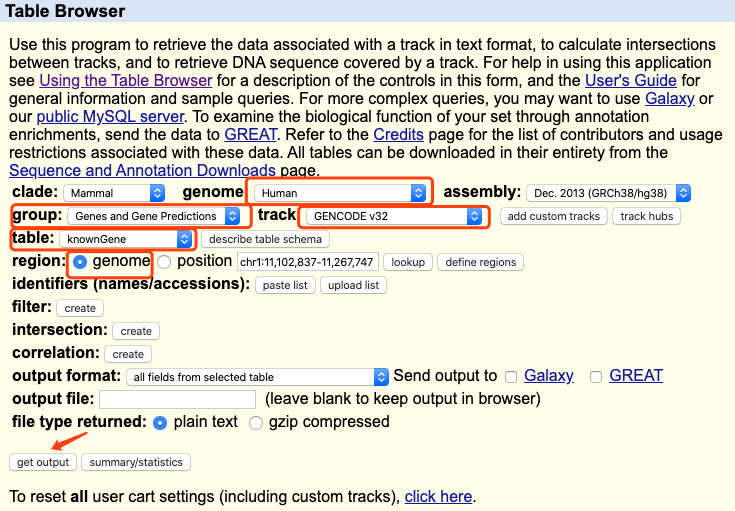
输出如下
1
2
3
4
5
6
7
8
9
#name chrom strand txStart txEnd cdsStart cdsEnd exonCount exonStarts exonEnds proteinID alignID
ENST00000456328.2 chr1 + 11868 14409 11868 11868 3 11868,12612,13220, 12227,12721,14409, uc286dmu.1
ENST00000450305.2 chr1 + 12009 13670 12009 12009 6 12009,12178,12612,12974,13220,13452, 12057,12227,12697,13052,13374,13670, uc286dmv.1
ENST00000488147.1 chr1 - 14403 29570 14403 14403 11 14403,15004,15795,16606,16857,17232,17605,17914,18267,24737,29533, 14501,15038,15947,16765,17055,17368,17742,18061,18366,24891,29570, uc286dmw.1
ENST00000619216.1 chr1 - 17368 17436 17368 17368 1 17368, 17436, uc031tla.1
ENST00000473358.1 chr1 + 29553 31097 29553 29553 3 29553,30563,30975, 30039,30667,31097, uc057aty.1
ENST00000469289.1 chr1 + 30266 31109 30266 30266 2 30266,30975, 30667,31109, uc057atz.1
ENST00000607096.1 chr1 + 30365 30503 30365 30365 1 30365, 30503, uc031tlb.1
注意
- 部分基因有多个TSS
- 以上输出的txStart就是TSS的位置
这样就得到了参考基因组的TSS位点。
2. 设定TSS区域并生成bed文件
得到TSS后,一般设定上下游1kb来作为TSS区域(即 txStart $\pm$ 1000bp,当然这个视具体情况设定),然后转化为TSS区域的bed文件。